Rapport technique final - Analyse du profil moléculaire des vaccins

Préface
Tout d'abord, nous tenons à remercier les commentaires très utiles de ceux qui ont examiné les résultats des analyses effectuées dans le cadre des activités de recherche scientifique relatives aux produits Priorix tetra et Infanrix Hexa. Les enjeux critiques présentés ont en effet été très utiles afin d'ajouter des intégrations technico-scientifiques permettant de clarifier le travail effectué. Nous pensons que ce n'est que par une saine communion de visions scientifiques que l'on pourra tirer des conclusions sur les données obtenues qui pourront être utiles à l'ensemble de la communauté scientifique et aux personnes qui les abordent.
1. Etat de l'art
Les études préliminaires (dépistage non soumis à confirmation) du profil biomoléculaire, métabolomique et protéomique, réalisées sur les produits Priorix Tetra et Infanrix Hexa ont conduit à un cadre de composition résumée dans les points suivants:
- Présence de différents signaux analytiques qui ne peuvent pas être associés à des composés connus grâce à la recherche sur les bases de données Metlin 1-2 et KEGG3. Par conséquent, une image est apparue associée à une complexité considérable dans la composition des produits commerciaux.
- Présence de protéines non déclarées dans une notice du produit Priorix Tetra. Ces derniers peuvent potentiellement être associés à des résidus du processus de production
- Non détection des antigènes déclarés dans le produit Infanrix Hexa. La technique d'analyse consistait en une digestion enzymatique avec de la trypsine associée à des techniques de spectrométrie de masse. 4-5
Ces données ont suscité plusieurs commentaires, en particulier concernant le point C - La détection des protéines est en effet réalisée selon une approche standard, internationalement reconnue depuis plus de 10 ans 4, de la digestion par l'enzyme trypsine 4. Les peptides ainsi obtenus sont séparés chromatographiquement et analysés par spectrométrie de masse 4-5. La principale observation est inhérente au fait que dans les vaccins, il existe des adjuvants à base d'aluminium qui pourraient potentiellement inhiber le processus de digestion enzymatique. Les données acquises par la suite ont permis d'apporter des éclaircissements substantiels, notamment en ce qui concerne la plainte exprimée au point C.
2. Nouvelles perspectives et analyses
2.1 Perspectives liées à l'analyse du produit Infanrix Hexa
Avant de procéder à l'illustration des nouvelles données acquises concernant les vaccins Hexyon et Gardas 9, il est essentiel de répondre à la question concernant le doute soulevé concernant l'inhibition de l'activité protéolytique de la trypsine causée par la présence d'adjuvants à base d'aluminium dans l'Infanrix Hexa vaccin. A cet égard, il faut préciser qu'un contrôle de la digestion est toujours présent au sein de la digestion tryptique. En fait, la trypsine utilisée pour effectuer la digestion, bien que conçue pour empêcher l'autolyse, a un petit pourcentage de cette dernière qui, dans le cas d'une activité enzymatique, conduit à l'obtention du fragment ayant m / z 842 et de la séquence peptidique suivante: VATVSLPR. Ce fragment a été effectivement détecté lors de la digestion tryptique du produit Infanrix Hexa comme vérifiable par le chromatogramme d'extraction ionique (figure 1).
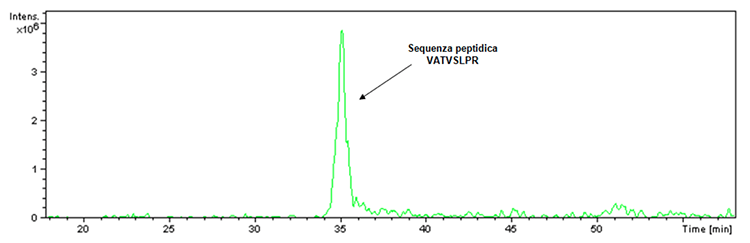
Figure 1: Chromatogramme d'extraction ionique associé aux ions avec un rapport m / z 842 trouvé dans l'échantillon du lot de produits Infanrix Hexa (lot n ° A21CD072D).
De plus, un contrôle externe est effectué par digestion de l'hémoglobine, afin de vérifier davantage la qualité du lot de trypsine utilisé. L'hémoglobine, analysée dans la section d'analyse dans laquelle le produit a été suivi, a été reconnue avec un score statistique significatif (loge <- 100). Ces données ont confirmé le fait que l'activité enzymatique était présente.
2.2 Nouvelles analyses concernant les produits Hexyon et Gardasil
L'analyse des produits Hexyon et Gardas 9 a permis de détecter des profils moléculaires complexes. Dans ce cas, cependant, la présence de la plupart des antigènes signalés dans la notice d'information a été détectée. Ils ont été détectés par digestion tryptique et en présence d'adjuvants.
Ce fait renforce encore la preuve que la réaction de digestion triptique n'est pas inhibée en présence d'adjuvants. Dans le cas des vaccins Hexyon et Gardas 9, la complexité du profil moléculaire a été principalement attribuée à la présence de nombreuses espèces, de faible poids moléculaire, non identifiables par les bases de données de référence Metlin 1-2- et KEGG 3.
3. Conclusions et considérations finales
Les analyses effectuées ont abouti à la conclusion suivante:
- Le profil moléculaire des vaccins analysés est généralement complexe et largement inconnu.
- Il existe des contaminations protéiques, non déclarées dans la notice, dont la composition est variable.
- Dans plusieurs cas, les antigènes déclarés dans la notice n'ont pas été détectés. Ce fait pourrait être attribué à plusieurs facteurs. Parmi ces derniers, on peut considérer la sensibilité de la méthode utilisée. Cependant, nous pensons pouvoir exclure le phénomène d'inhibition de la digestion dû à la présence d'adjuvants dans la formulation du vaccin. En effet, l'activité enzymatique est principalement confirmée par la présence de fragments d'autolyse tryptique, au sein des solutions des vaccins digérés (contrôle interne).
4. Études futures
D'autres études seront menées dans le cadre des activités de recherche et développement visant à étudier les aspects suivants:
- composition macromoléculaire associée aux résidus solides présents dans les vaccins (analyse MALDI TOF MS); 5
- évaluation de la concentration des métaux présents dans les produits.
- Analyse de deuxième niveau pour confirmer la présence de composés toxiques détectés en phase de criblage. Leur concentration sera alors liée à leur toxicité selon ce qui est indiqué sur les fiches de données de sécurité internationales. Les analyses de second niveau seront réalisées conformément à la directive européenne UE 2002/657 / CE, utile pour garantir des normes de qualité élevées dans le secteur de la spectrométrie de masse. 6
Loretta Bolgan
5. Références bibliographiques
- Autenhahn R, Cho K, Uritboonthai W, Zhu Z, Patti G, Siuzdak G (septembre 2012). "Un flux de travail accéléré pour la métabolomique non ciblée utilisant la base de données METLIN". Biotechnologie de la nature. 30: 826–828. doi: 10.1038 / nbt.2348. PMC 3666346. PMID 22965049.
- Smith CA, I'Maille G, Want EJ, Qin C, Trauger SA, Brandon TR, Custodio DE, Abagyan R, Siuzdak G (décembre 2005). "METLIN: une base de données spectrale de masse des métabolites" (PDF). Ther Drug Monit. 27 (6): 747–51. doi: 10.1097 / 01.ftd.0000179845.53213.39. PMID 16404815.
- Kanehisa M (2013). "Evolution chimique et génomique des réseaux de réaction catalysés par des enzymes". FEBS Lett.587 (17): 2731–7.
- Cristoni S, Bernardi LR. "Bioinformatique dans l'analyse des données de spectrométrie de masse pour les études de protéomique." Expert Rev Proteomics. 2004 déc; 1 (4): 469-83.
- Cristoni S, Bernardi LR. "Développement de nouvelles méthodologies pour l'étude par spectrométrie de masse des macromolécules bioorganiques." Mass Spectrom Rev.2003 Nov-Dec; 22 (6): 369-406.
- Cristoni S, Dusi G, Brambilla P, Albini A, Conti M, Brambilla M, Bruno A, Di Gaudio F, Ferlin L, Tazzari V, Mengozzi S, Barera S, Sialer C, Trenti T, Cantu M, Rossi Bernardi L, Noonan DM. "SANIST: optimisation d'une technologie d'identification des composés basée sur la directive de l'Union européenne avec des applications en analyses médico-légales, pharmaceutiques et alimentaires." J. Mass Spectrom. Jan 2017; 52 (1): 16-21. doi: 10.1002 / jms.3895.
Télécharger : CORVELVA-finale-technique-Report.pdf
Traduit par l'équipe CLiVa - www.clivatoscana.com

