Presentazione in breve dei risultati
Vi presentiamo oggi un ulteriore importantissimo approfondimento sul sequenziamento genomico della linea cellulare riscontrata all'interno del vaccino quadrivalente MPRV (morbillo, rosolia, parotite e varicella) Priorix Tetra, e quello della linea cellulare MRC-5 (che è quella dichiaratamente utilizzata nello sviluppo del vaccino medesimo).
Riassumendo i precedenti report pubblicati:
- abbiamo dapprima dimostrato come il quantitativo di DNA presente nel vaccino summenzionato fosse ben al di sopra del consentito;
- abbiamo poi verificato che la linea cellulare è effettivamente la MRC-5 dichiarata;
- abbiamo successivamente proceduto al sequenziamento del genoma della linea cellulare estratta direttamente dal vaccino, evidenziando mutazioni pesantissime che comportano la possibilità di gravi rischi per la salute nell'utilizzo di questo vaccino.
Quello che qui Vi presentiamo è appunto il lavoro di comparazione tra i due genomi: quello che realmente abbiamo trovato all'interno del vaccino e quello della MRC-5 acquistata presso la banca dati ATCC.
Il risultato è che le due linee presentano importanti differenze dal punto di vista della stabilità genetica. In particolare il genoma vaccinale risulta essere pesantemente modificato rispetto a quello della linea cellulare depositata nel 1966.
Ciò significa che i produttori, in questo caso del Priorix tetra, hanno acquistato la linea cellulare in condizioni ottimali dal punto di vista della stabilità genetica e con l’uso continuativo nel tempo a scopo di produzione hanno messo in commercio vaccini contenenti materiale genetico umano progressivamente sempre più modificato e pericoloso per la salute dei vaccinati.
Poiché le agenzie regolatorie non richiedono il controllo periodico della stabilità genetica delle linee fetali e non pongono nemmeno una soglia minima di sicurezza, ne possiamo dedurre che non è stato rispettato già da diverso tempo un requisito fondamentale per la qualità e la sicurezza dei vaccini che vengono ottenuti da linee cellulari umane e cioè la necessità di verificare che non acquisiscano mutazioni e riarrangiamenti potenzialmente pericolosi e di garantire l’eliminazione dei residui di lavorazione di materiale genetico qualora presenti.
Non sta a noi identificare quale parte del processo produttivo o conservativo possa inficiare la qualità e la sicurezza del prodotto ma quello che oggi possiamo testimoniare pubblicando questo report è che urge da parte degli enti di controllo un approfondito esame dei risultati che le nostre analisi hanno evidenziato in questi due anni, focalizzando l'attenzione sul prodotto finito, venduto e somministrato e non soltanto sui singoli “ingredienti” autorizzati.
Ricordiamo ancora una volta che questi prodotti fanno parte del calendario vaccinale italiano e vengono attualmente obbligatoriamente somministrati alla popolazione pediatrica del nostro Paese nonché di altri paesi europei e in generale in tutto il mondo.
Gli enti di controllo hanno una responsabilità enorme rispetto alla sicurezza che viene attualmente sbandierata a parole ma che nei fatti, a quanto pare, e a quanto risulta da queste nostre analisi, non può essere garantita ed è anzi evidentemente disattesa.
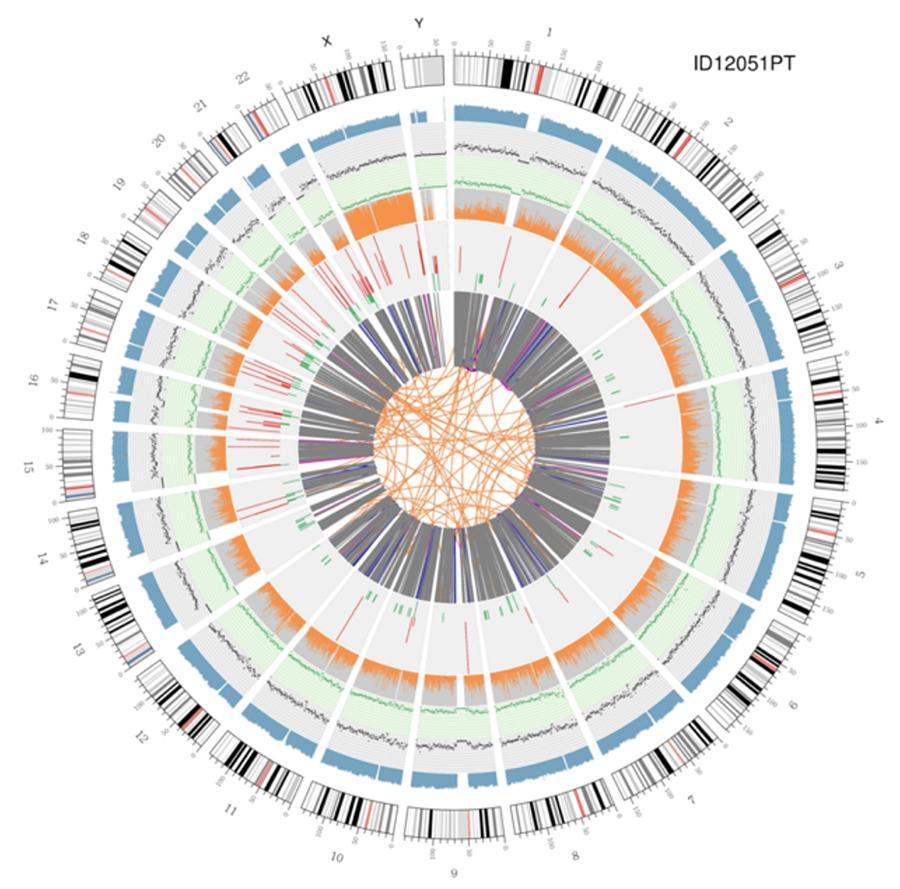
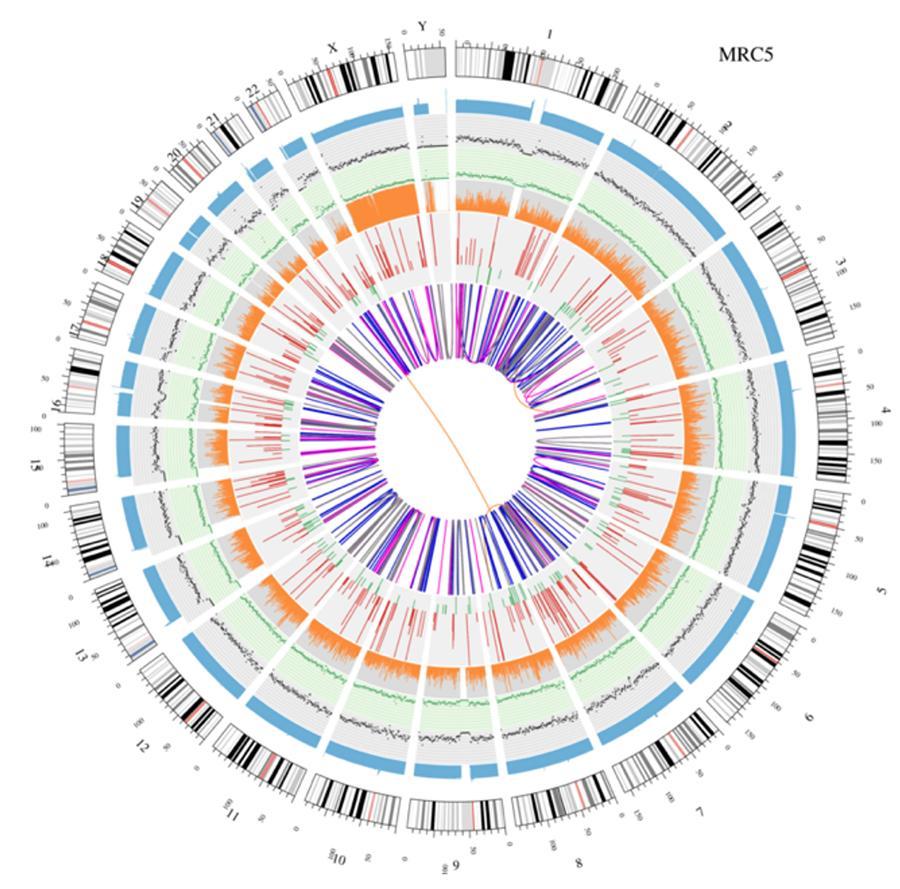
Visualizzazione circolare dei genomi (circos plot)
Una rappresentazione grafica dei due genomi chiamata ‘circos plot’ è riportata di seguito.
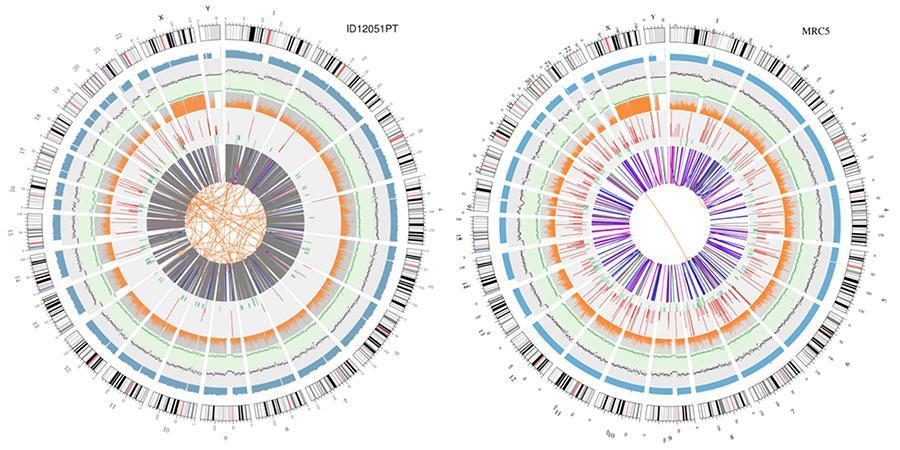
Significato dei vari cerchi concentrici
- Il cerchio esterno (il primo cerchio) è il numero del cromosoma.
- Il secondo anello (azzurro) rappresenta la copertura di reads in stile istogramma. Ogni istogramma è la copertura media di un'area di 0,5 Mbp.
- Il terzo anello (nero) rappresenta la densità delle INDELs in stile ‘grafico di dispersione’. Ogni punto nero viene calcolato come numero di INDELs (Inserzioni/delezioni di piccole dimensioni) in un intervallo di 1 Mbp.
- Il quarto anello (verde) rappresenta la densità snp in stile ‘grafico di dispersione’. Ogni punto verde viene calcolato come numero di SNPs (varianti a singolo nucleotide) in un intervallo di 1 Mbp.
- Il quinto anello rappresenta la proporzione di SNP in omozigosi (arancione) e in eterozigosi (grigio) in stile istogramma. Ogni istogramma viene calcolato da una regione 1 Mbp.
- Il sesto anello rappresenta l'inferenza di CNV (Varianti in numero di copie). Il rosso significa guadagno di pezzi di DNA e il verde significa perdita.
- L'anello più centrale rappresenta l'inferenza di SV (Varianti Strutturali) nelle regioni esoniche e di splicing. TRA (arancione, traslocazioni), INS (verde, inserzioni), DEL (delezioni, grigio), DUP (duplicazioni, rosa) e INV (inversioni, blu).
Conclusioni
I due genomi mostrano importanti differenze. In particolare, il genoma vaccinale risulta essere pesantemente mutato rispetto a quello della linea cellulare depositata nel 1966. Va detto che non abbiamo avuto la possibilità di sequenziare la linea cellulare che viene utilizzata dalla GlaxoSmithKline per la produzione del vaccino Priorix Tetra, ma quella dello stock originale. Le aziende farmaceutiche che utilizzano linee cellulari per la produzione di farmaci e vaccini, hanno la loro linea MRC-5 ma questo non cambia i nostri sospetti, ovvero il fatto che non venga richiesto periodicamente il controllo della stabilità genetica e questo è gravissimo a nostro avviso, soprattutto perché ne è consentita la presenza senza soglia limite.
Download: CORVELVA-Comparazione-genoma-umano-Priorix-Tetra-e-linea-MRC-5.pdf
Comparazione tra genoma umano contenuto in Priorix Tetra e nella linea cellulare MRC-5 (ATCC standard MRC-5 ATCC® CCL-171™)
La presenza del virus della rosolia è stata dimostrata sequenziando una libreria RNA-seq ad altissima profondità (circa 260 milioni di sequenze Illumina prodotte). Sono state rilevate 114 sequenze su 260 milioni, pari allo 0.00004% del totale delle sequenze. Le sequenze del genoma della rosolia sono state quindi confermate manualmente con il software BLAST (Basic Local Alignment Search Tool, https://blast.ncbi.nlm.nih.gov/Blast.cgi). La libreria sequenziata a più bassa profondità (circa 12 milioni di sequenze accoppiate Illumina, pari a 6 milioni di frammenti di libreria sequenziati) non aveva rilevato la presenza di NESSUNA reads attribuibile alla rosolia, in questo lotto.
Anche le sequenze degli altri virus presenti nel vaccino (varicella, morbillo e parotite) sono state validate con la stessa modalità manuale, confermando di essere state correttamente assegnate.
Introduzione
I sequenziatori di nuova generazione sono diventati strumenti d’elezione per analisi approfondite nel campo della biologia e della medicina, soprattutto quella di precisione. Questi strumenti consentono di approcciarsi in maniera nuova e più globale ad una serie di applicazioni come sequenziamento de novo, studi di metagenomica, di epigenomica, sequenziamento del trascrittoma e risequenziamento di genomi.
Quest’ultima applicazione (risequenziamento) è molto diffusa in campo umano sia a scopo ricerca che diagnostico e consiste nel sequenziamento con tecnologia NGS (Next Generation Sequencing) di un intero genoma individuale allo scopo di mappare mutazioni di singolo nucleotide (SNP, pronuncia ‘snip’), inserzioni e delezioni di sequenze più o meno lunghe avvenute in determinate posizioni del genoma e variazioni nel numero di copie di porzioni di genoma/geni (CNV, Copy Number Variants).
La procedura di sequenziamento prevede che il DNA di un individuo venga spezzato meccanicamente in frammenti di piccole dimensioni (400-500 paia di basi) e ai frammenti vengano legate dei tratti di DNA artificiale chiamati adattatori, che permettono di legare i frammenti di DNA umano ad una superficie di vetro sulla quale si esegue poi la lettura delle basi (A, C, G, T). La lettura delle basi del DNA avviene mediante reazioni chimiche di incorporazione di nucleotidi marcati con molecole fluorescenti. I milioni di sequenze (reads) che si ottengono dal sequenziamento avvenuto sulla superficie di vetro, vengono poi mappate sul genoma di riferimento umano con opportuni software e quindi identificate tutte le varianti presenti nel genoma analizzato rispetto al riferimento.
Questa stessa procedura è stata eseguita sul genoma umano presente in Priorix Tetra lot. n. A71CB256A e sul DNA estratto dalla linea cellulare MRC-5, depositata nel 1966 presso ATCC (MRC-5 ATCC® CCL-171™).
ATCC è la principale organizzazione a livello mondiale di risorse e standard di materiali biologici la cui missione si concentra sull'acquisizione, autenticazione, produzione, conservazione, sviluppo e distribuzione di microrganismi di riferimento, linee cellulari e altri materiali biologici standard. Fu istituita nel 1925, quando un comitato di scienziati riconobbe la necessità di una raccolta centralizzata di materiali biologici che potesse servire gli scienziati di tutto il mondo.
La linea cellulare MRC-5 deriva da un tessuto polmonare normale di un feto maschio di 14 settimane ed è stata depositata in ATCC da J.P. Jacobs nel settembre del 1966. Queste cellule sono dichiarate in grado di raddoppiare da 42 a 46 popolazioni prima dell'inizio della senescenza. L’articolo di riferimento per questa linea cellulare risale al 1970: https://www.nature.com/articles/227168a0
Risultati
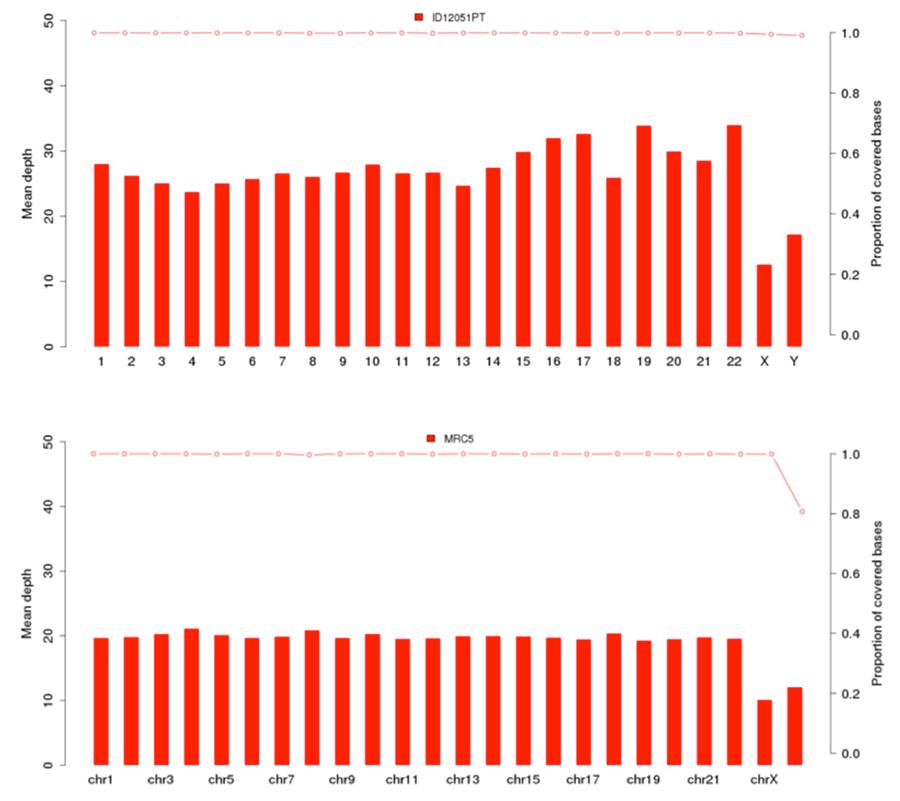
Come già precedentemente mostrato, Il DNA umano presente in Priorix Tetra lot. n. A71CB256A (indicato nei grafici seguenti come campione ID12051PT) è un genoma individuale completo ovvero è presente DNA genomico di tutti i cromosomi di un individuo di sesso maschile. Di seguito grafici di copertura (Mean depth= copertura media) dei 22 cromosomi umani e dei cromosomi X e Y, per il vaccino (immagine sopra) e la linea cellulare MRC5 (immagine sotto).
Di seguito vengono riportati i risultati dell’analisi dei vari tipi di varianti.
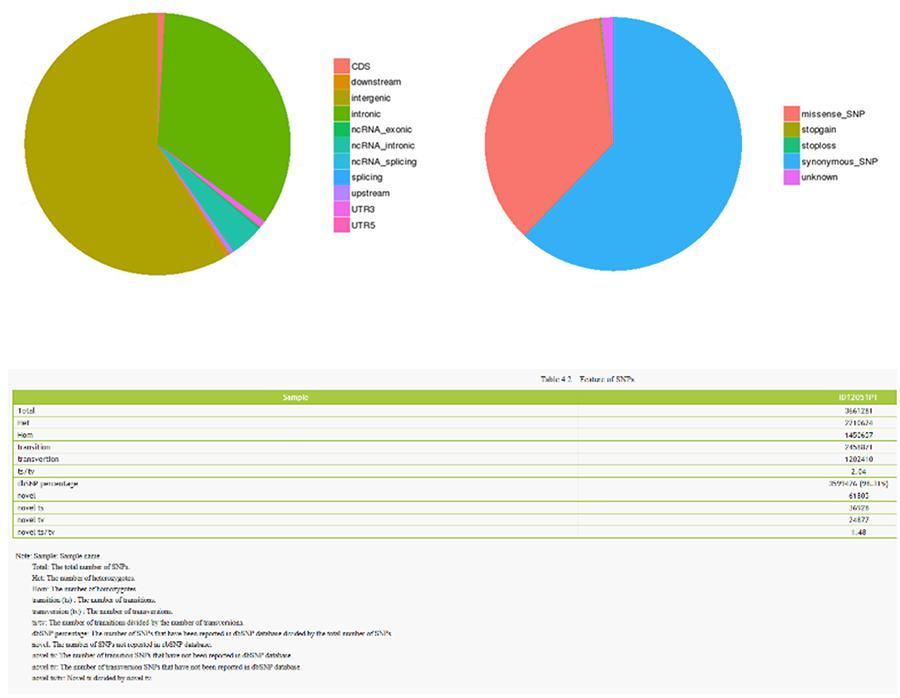
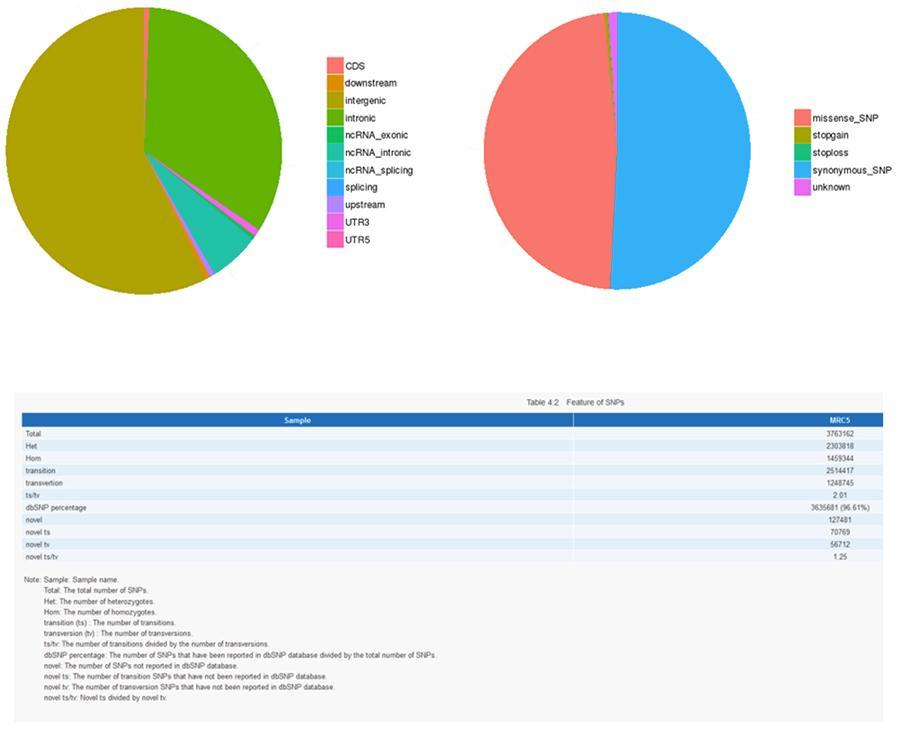
Varianti a singolo nucleotide (SNP) e corte inserzioni/delezioni (InDels)
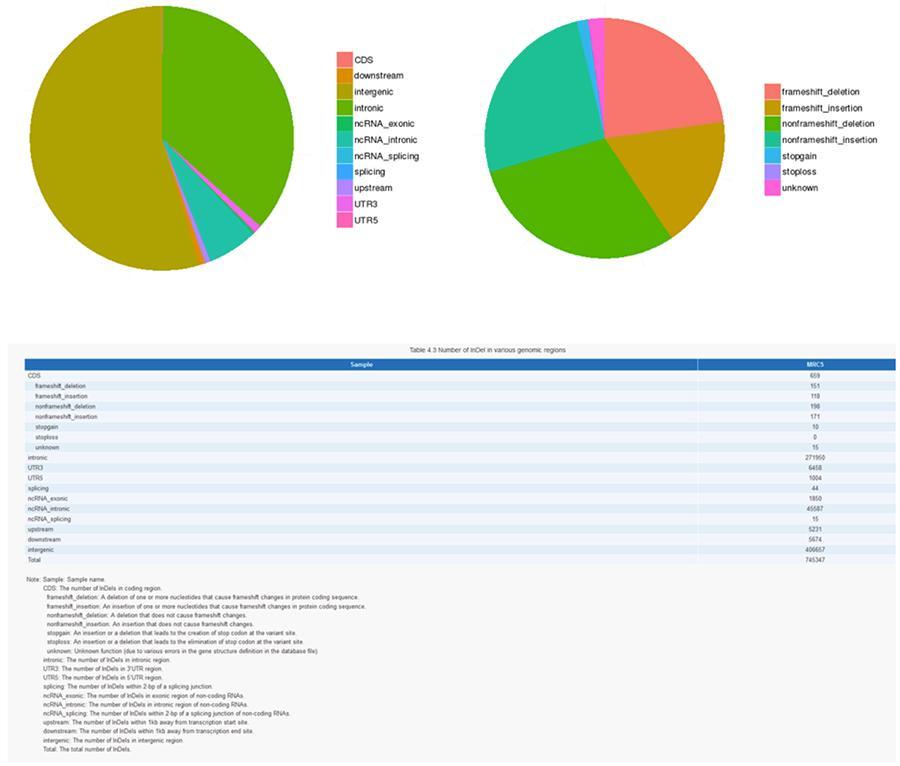
Le varianti di singole basi del DNA (SNP, pronuncia ‘snip’) sono polimorfismi, cioè variazioni del materiale genetico, a carico di un unico nucleotide. Le ‘InDels’ sono invece piccole inserzioni e delezioni di meno di 50pb di lunghezza e costituiscono un'altra classe di varianti genomiche nel genoma umano.
SNP (varianti a singolo nucleotide) - ID12051PT
SNP (varianti a singolo nucleotide) - MRC-5
INDELs (piccole inserzioni/delezioni) - ID12051PT
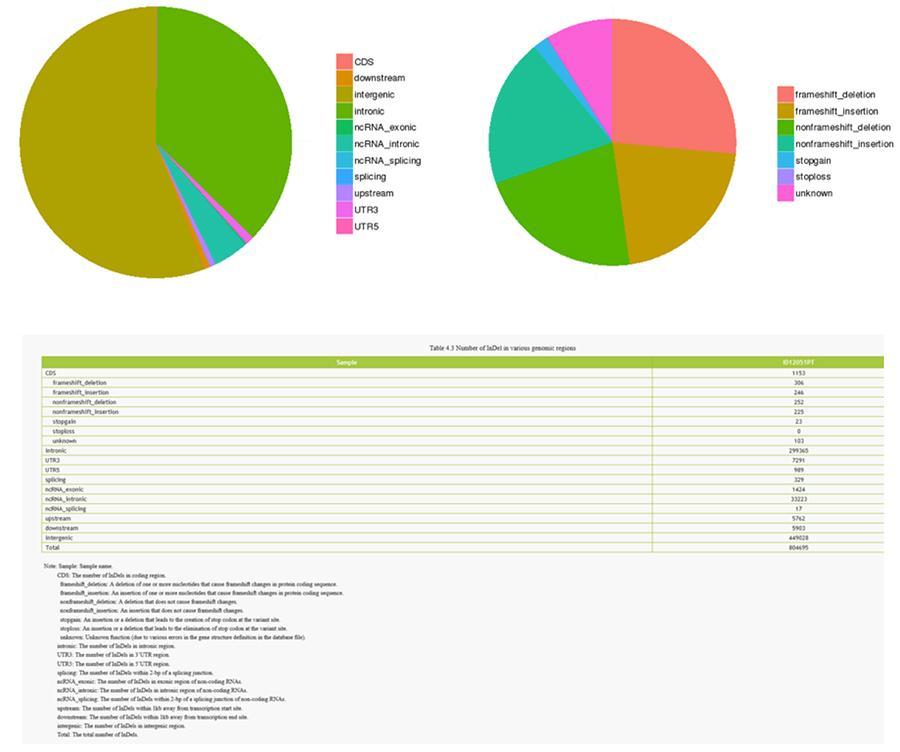
INDELs (piccole inserzioni/delezioni) - MRC-5
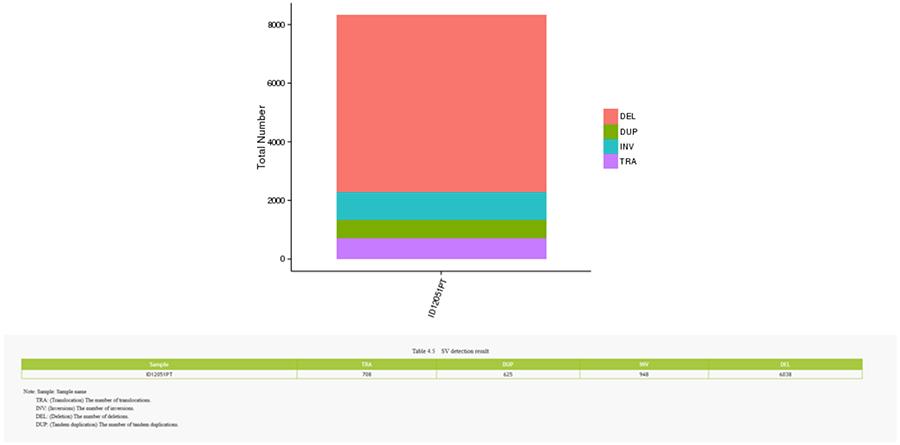
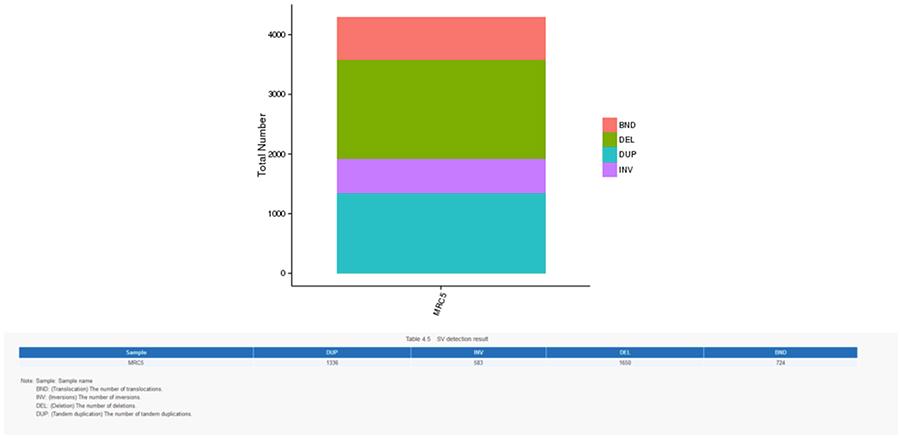
CNV (Copy Number Variants) e SV (Structural Variants)
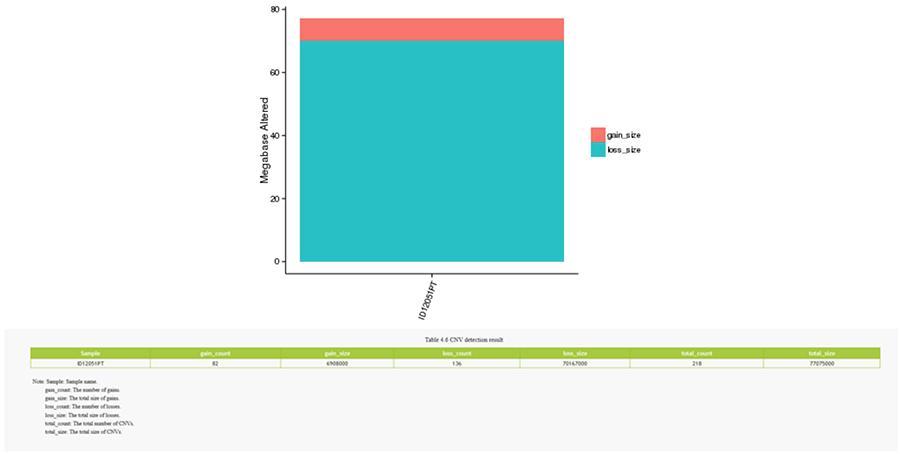
Le varianti in numero di copie (CNV) sono varianti genomiche dovute a variazioni nel numero di copie di frammenti relativamente grandi (più lunghi di 50 bp) tra genomi individuali. Esistono due tipi di CNV: tipo ‘gain’ (guadagno di copie) e tipo ‘loss’ (perdita di copie).
Le varianti strutturali (SV) sono varianti genomiche con dimensioni relativamente grandi (> 50 bp), tra cui delezioni, duplicazioni, inserzioni, inversioni e traslocazioni.
CNV (varianti in numero di copie)
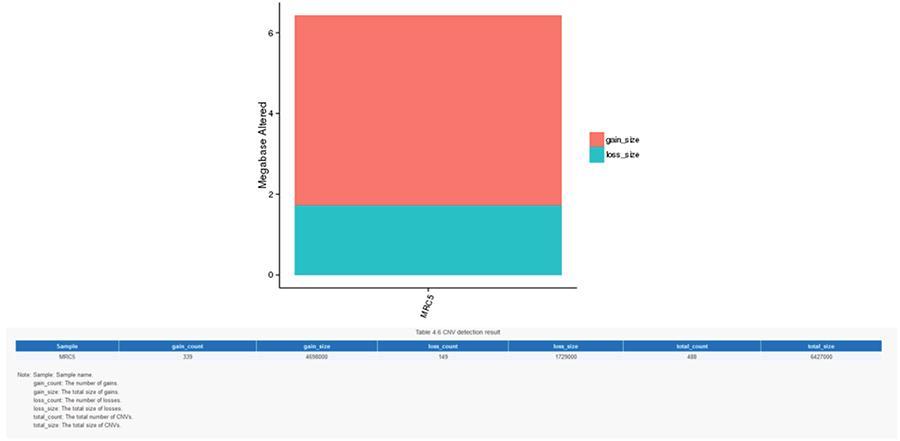
SV (varianti strutturali)
Visualizzazione circolare dei genomi (circos plot)
Una rappresentazione grafica dei due genomi chiamata ‘circos plot’ è riportata di seguito.
Significato dei vari cerchi concentrici
- Il cerchio esterno (il primo cerchio) è il numero del cromosoma.
- Il secondo anello (azzurro) rappresenta la copertura di reads in stile istogramma. Ogni istogramma è la copertura media di un'area di 0,5 Mbp.
- Il terzo anello (nero) rappresenta la densità delle INDELs in stile ‘grafico di dispersione’. Ogni punto nero viene calcolato come numero di INDELs (Inserzioni/delezioni di piccole dimensioni) in un intervallo di 1 Mbp.
- Il quarto anello (verde) rappresenta la densità snp in stile ‘grafico di dispersione’. Ogni punto verde viene calcolato come numero di SNPs (varianti a singolo nucleotide) in un intervallo di 1 Mbp.
- Il quinto anello rappresenta la proporzione di SNP in omozigosi (arancione) e in eterozigosi (grigio) in stile istogramma. Ogni istogramma viene calcolato da una regione 1 Mbp.
- Il sesto anello rappresenta l'inferenza di CNV (Varianti in numero di copie). Il rosso significa guadagno di pezzi di DNA e il verde significa perdita.
- L'anello più centrale rappresenta l'inferenza di SV (Varianti Strutturali) nelle regioni esoniche e di splicing. TRA (arancione, traslocazioni), INS (verde, inserzioni), DEL (delezioni, grigio), DUP (duplicazioni, rosa) e INV (inversioni, blu).
Conclusioni
I due genomi mostrano importanti differenze. In particolare, il genoma vaccinale risulta essere pesantemente mutato rispetto a quello della linea cellulare depositata nel 1966. Va detto che non abbiamo avuto la possibilità di sequenziare la linea cellulare che viene utilizzata dalla GlaxoSmithKline per la produzione del vaccino Priorix Tetra, ma quella dello stock originale. Le aziende farmaceutiche che utilizzano linee cellulari per la produzione di farmaci e vaccini, hanno la loro linea MRC-5 ma questo non cambia i nostri sospetti, ovvero il fatto che non venga richiesto periodicamente il controllo della stabilità genetica e questo è gravissimo a nostro avviso, soprattutto perché ne è consentita la presenza senza soglia limite.



